El Virus de la Necrosis Hipodérmica y Hematopoyética Infecciosa está establecido en estanques camaroneros en Ecuador

En los últimos años, el Virus de la Necrosis Hipodérmica y Hematopoyética Infecciosa (IHHNV), un patógeno del camarón incluido en la lista de la Organización Mundial de Sanidad Animal [WOAH; fundada como la Oficina Internacional de Epizootias, OIE] ha sido reportada como la causa del crecimiento deficiente en el camarón tigre negro (Penaeus monodon) y el camarón blanco del Pacífico (Penaeus vannamei) por varias publicaciones en Australia, India y, más recientemente, Ecuador.
El tema que pretendemos explorar aquí es: “¿Cuál es el patrón del virus IHHN, los elementos virales endógenos del IHHNV (EVEs, secuencias de ADN derivadas de un virus y presentes en la línea germinal de un organismo no viral) y los camarones con resultado negativo en la prueba PCR de IHHN en estanques camaroneros en Ecuador, y qué implican estos patrones con respecto a la tolerancia a este virus en granjas camaroneras en Ecuador.”
El IHHNV, introducido por primera vez a mediados de la década de 1980 a Ecuador a través de P. monodon importado de Asia, está bien establecido y es autóctono en camarones silvestres en las Américas desde Perú hasta el extremo norte del Golfo de California, además de ser reportado en camarones silvestres recolectados en el Golfo de México.
En términos evolutivos, los crustáceos aparecieron por primera vez hace unos 455 millones de años. La familia de camarones Penaeidae, como miembros de Dendrobranchiata (un suborden de decápodos, comúnmente conocidos como langostinos), representan ejemplos de eones de adaptación evolutiva exitosa a través de mecanismos mediados por generaciones. Aparentemente, uno de esos mecanismos es la incorporación de secuencias de ADN que reflejan las secuencias de patógenos dentro del genoma del huésped, lo que lleva a estrategias de defensa mediadas por el ARN del huésped.
La resiliencia natural del camarón ha sido reconocida durante mucho tiempo por los productores de camarón y, en particular, por los productores de L. vannamei en América Latina, donde su capacidad para adaptarse a los patógenos se refleja en el uso de la estrategia de manejo conocida por el acrónimo APE (All Pathogens Exposed, o Expuesto a Todos los Patógenos). Además, también está bien establecida la tolerancia a patógenos de camarones seleccionados mediante la cría selectiva de poblaciones de camarones libres de patógenos específicos (SPF) en empresas modernas de genética de camarones.
Durante la última década, varios investigadores – particularmente en Tailandia bajo la dirección del Prof. T.W. Flegel y por la Dra. V. Alday-Sanz y asociados en otros lugares – han documentado la mecánica de los procesos involucrados a nivel molecular y han demostrado la utilidad de la selección de camarones SPF mediante pruebas en animales individuales para establecer L. vannamei tolerantes a, y sin patógenos derivados de Ecuador y transferidos a otras áreas del mundo. Sus trabajos han sido ampliamente documentados y se erigen como una importante contribución fundamental para la comprensión de cómo estos crustáceos económicamente importantes han evolucionado y prosperado.
Aplicando la metodología de PCR estándar utilizando cebadores de PCR listados por la OIE, en este artículo reportamos los patrones de distribución – medidos con métodos de PCR estándar – de secuencias de ADN de IHHNV insertadas (EVEs), infección real (probable) de IHHNV y camarones negativos a IHHNV por prueba de PCR recolectados en estanques en Ecuador en 2021. También discutimos la relevancia de los hallazgos de laboratorio, ya que estos se relacionan con el estado del diagnóstico de IHHNV en camarones de cultivo en esta región. Estos resultados respaldan la conclusión de que el IHHNV no es un patógeno significativo dentro de los acervos genéticos de L. vannamei examinados en nuestro estudio. Además, nuestros datos indican que los cebadores y la metodología de PCR enumerados por la OIE actualmente aplicados están desactualizados debido a la presencia de EVEs en los camarones cultivados.
Un artículo reciente sobre IHHNV en Ecuador destacó brevemente la tolerancia demostrada por una mínima patología tisular inducida por el virus IHHN en camarones infectados con el virus IHHN, al mismo tiempo que reportaba un alto título de IHHNV en las muestras de tejido. Fundamentalmente hablando, estas dos interpretaciones no son compatibles, ya que la replicación del virus IHHN dentro de las células produce alteraciones anatómicas fácilmente visibles en secciones histológicas de tejido teñidas.
El propósito de nuestro artículo es proporcionar una interpretación diferente basada en la presencia de EVEs de IHHNV, así como proporcionar información adicional para reforzar que, a lo largo del tiempo generacional, los camarones y los patógenos que los afectan modifican continuamente sus relaciones huésped/patógeno, todo parte integrante de los altamente mecanismos de defensa y autopreservación desarrollados dentro de una población de camarones.
Configuración del estudio
Se recolectaron muestras de camarones de 134 estanques (33 de vivero y 101 de engorde) en granjas a lo largo de la costa del Pacífico de Ecuador en 2021. Los camarones en las muestras de los estanques de engorde se pesaron hasta 0,1 gramos, se diseccionaron y se extrajo tejido branquial/pleópodo, que fue preservado en etanol al 95 por ciento de cada animal. Las muestras de estanques de crianza no se pesaron cuando se recolectaron muestras de tejido. Las pruebas PCR fueron realizadas por personal de la unidad de I+D de la empresa camaronera ecuatoriana Empagran S.A.
Cada muestra se extrajo y analizó utilizando los cebadores de PCR 309F/R, 389F/R, 392F/R 77012/77353, MG831F/R de acuerdo con los métodos enumerados por la OIE. Estos cebadores son los que se enumeran en el Manual Acuático de la OIE (OIE Aquatic Manual) para la detección del HHNV por PCR estándar para el IHHNV. No se realizaron análisis de secuencia en ninguna de las muestras de este estudio.
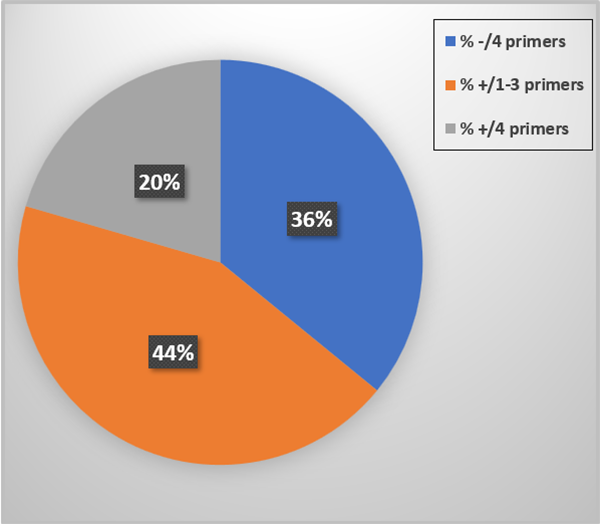
Los resultados de la prueba de PCR se resumen en la Fig. 1. El 36 por ciento de las muestras de tejido no se cebaron con ninguno de los conjuntos de cebadores y el 44 por ciento reaccionó con uno-tres de los cuatro conjuntos de cebadores. Estos representan EVEs, secuencias de ADN incrustadas en el ADN genómico del huésped. Finalmente, el 20 por ciento dio positivo en la prueba de PCR para los cuatro conjuntos de cebadores aplicados. Dado que los EVEs y el IHHNV vivo pueden ocurrir juntos en el mismo camarón, interpretamos que estos animales son “sospechosos” de que la prueba de PCR sea positiva para el IHHNV (virus vivo). Ninguna de las muestras de tejido reaccionó con los cebadores MG831F/R.
Los EVEs son secuencias de ADN del exterior incrustadas en el ADN del camarón huésped. Como tal, los EVEs pasan de una generación de camarones a su descendencia de acuerdo con los Principios de la Genética Mendeliana. Como los EVEs se encuentran dentro del ADN de cada célula del cuerpo de un camarón, predecimos que las pruebas de PCR cuantitativas arrojarán un Cq (Valor de Cuantificación) bajo, una indicación de que la secuencia de ADN objetivo es abundante en la muestra. Si no se tiene en cuenta la posibilidad de que un EVE esté impulsando la reacción de qPCR, entonces el valor bajo de Cq puede malinterpretarse para indicar que la señal refleja una abundancia de virus vivo en una muestra cuando el “título alto” también podría ser tan bien explicado por la presencia de un EVE y/o si hubiera un virus vivo presente, junto con un EVE, su presencia podría ser baja pero parece alta debido a la contribución del EVE a la secuencia general que reacciona en el protocolo qPCR.
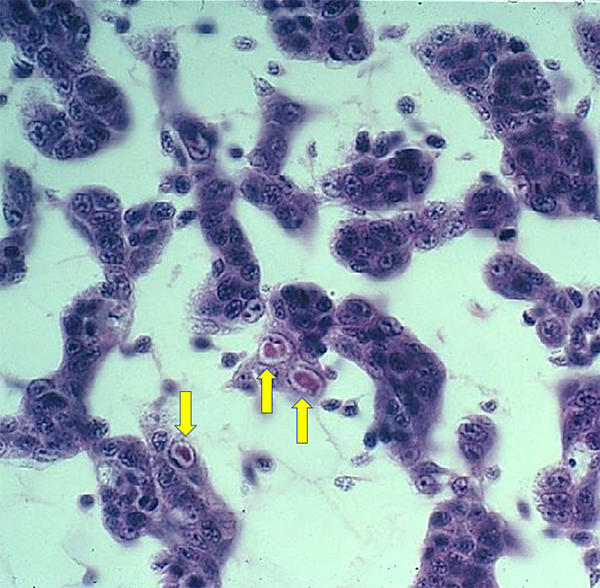
Como se mostró anteriormente, el IHHNV vivo en muestras de tejido de camarón – cuando se analiza con múltiples cebadores de PCR – se cebará con todos los cebadores porque todos deben estar presentes para que un virus funcional se replique. Mientras que, si al usar un panel de cebadores, si algunos pero no todos los cebadores se amplifican, este patrón es una clara indicación de que los cebadores de PCR están midiendo un EVE y no el virus completo. Además, la replicación del IHHNV da como resultado alteraciones anatómicas dentro de los núcleos de las células objetivo visibles con tinción H&E (ver Fig. 2) o métodos de sonda génica. Un título alto de virus vivo irá acompañado de una gran abundancia de células huésped anatómicamente alteradas, y los síntomas de la enfermedad del IHHNV están relacionados con la abundancia de células huésped infectadas, anatómicamente alteradas que pierden su función normal.
La distribución de PCR negativos, EVE positivos (1-3 cebadores) y sospechosos de IHHNV (4 cebadores) dentro de agrupaciones organizadas por peso de camarón dentro de un rango de tamaño se resumen en la Tabla 1.
Brock, IHHNV, Tabla 1
| Cebador | Cebador | Cebador | Cebador | Promedio | |
|---|---|---|---|---|---|
| Grupo de peso | 309 | 392 | 389 | 77012 | – |
| <1g | 1 | 0.83 | 1 | 0.65 | 0.87 |
| 1-5g | 1 | 0.83 | 1 | 0.44 | 0.82 |
| 5-10g | 0.79 | 1 | 1 | 1 | 0.81 |
| >10g | 1 | 0.78 | 1 | 0.46 | 0.81 |
Los cálculos anteriores se realizaron eliminando todos los camarones reactores de cebador sin PCR para considerar solo aquellos animales cuya muestra de tejido se cebó con uno o más de los cuatro conjuntos de cebadores. Dentro del subconjunto de camarones reactores primarios de IHHNV, organizados por grupos de peso, en promedio para los cuatro grupos de tamaño, la prevalencia de reactores positivos fue bastante similar (+/- ~6 por ciento) entre los cuatro grupos de tamaño. Los datos organizados de esta manera no respaldan la conclusión de una relación positiva obvia entre el estado positivo del cebador de PCR frente al agrupamiento por peso de los camarones.
El Manual Acuático de la OIE (Capítulo 2.2.4. – Infección por el Virus de la Necrosis Hipodérmica y Hematopoyética Infecciosa, Sección 7. Los criterios de diagnóstico corroborativo brindan orientación para discriminar entre un caso sospechoso y un caso confirmado) proporciona los puntos de referencia que se citan a continuación:
7.1. Definición de caso sospechoso: Se sospecha de infección por IHHNV si se cumple al menos uno de los siguientes criterios: i) Signos clínicos indicativos de infección por IHHNV o ii) Histopatología indicativa de infección por IHHNV o iii) Resultado positivo por PCR.
7.2. Definición de caso confirmado: La infección por el IHHNV se confirma si se cumplen dos de los siguientes criterios: i) Resultado positivo por hibridación in situ; ii) Resultado positivo por PCR (siempre específico de genotipo); y iii) Análisis de secuencia para confirmar la secuencia de ácido nucleico de IHHNV.
Estamos de acuerdo con otros en que los protocolos moleculares actuales de la OIE para IHHNV están desactualizados desde la perspectiva de los EVEs, y que se necesitan cambios para evitar diagnósticos de etiología incorrectos adicionales, ya que esto se relaciona con la enfermedad de IHHNV en camarones peneidos cultivados. Aunque hay disponibles publicaciones científicas de calidad revisadas por pares sobre los EVEs y mecanismos de tolerancia propuestos a nivel molecular, las metodologías diseñadas hace años y adoptadas en el Manual Acuático de la OIE para el IHHNV para abordar el problema de los EVEs son inadecuadas hoy en día y conducen a diagnósticos erróneos de la actual importancia del IHHNV para la salud del camarón.
Por ejemplo, la OIE establece: “En caso de que los resultados sean ambiguos con el juego de cebadores “universales” 389F/R, se recomienda usar cebadores de una región diferente del genoma para las pruebas de confirmación. En este caso, eso significaría conjuntos de cebadores 77013/77353 o 392F/R para confirmación.” Con base en los resultados de nuestro estudio, esta afirmación ya no es correcta y recomendamos una revisión de los protocolos.
El conjunto de cebadores MG831F/R no se detectó en ninguna de las 134 muestras de tejido analizadas en este estudio. Basado en estos hallazgos, no se demostró que los cebadores MG831F/R fueran útiles para la detección del IHHNV en L. vannamei.
Perspectivas
Como los EVEs están dentro del genoma del huésped y el genoma del huésped está dentro de cada célula del cuerpo del camarón, se esperaría que un EVE o EVEs, si están presentes y medidos, produjera un valor de Cq bajo y, por lo tanto, se interpreten como una muestra de tejido con título alto de virus vivo en el tejido del huésped en escenarios donde los EVEs no están presentes. Sin embargo, si existen EVEs entonces estas fuentes de secuencias de ADN con el genoma del huésped también deben tenerse en cuenta como una posible explicación alternativa. Los datos de PCR reportados aquí son un buen ejemplo de la validez de que esto es correcto.
Por lo tanto, medir el IHHNV asociado con camarones que muestran un crecimiento lento sin establecer que los camarones involucrados también tienen una alta frecuencia de células infectadas con IHHNV en los órganos diana, o realizar análisis de secuencia para confirmar la presencia del genoma del virus trae consigo un nivel de incertidumbre y puede resultar en un diagnóstico etiológico incorrecto. Aunque existe confusión con la interpretación debido a los EVEs con los cebadores de PCR del IHNNV de la OIE actualmente enumerados, también hay desinformación porque los diagnosticadores y los investigadores pueden no seguir los estándares de la OIE en lo que respecta a un caso sospechoso frente a un caso confirmado porque no logran realizar los pasos de confirmación de la OIE para validar sus resultados de estado sospechoso de IHHNV.
Actualmente, el IHHNV aún es un patógeno incluido en la lista de la OIE y la importación de camarones vivos no es permitida por las autoridades de entrada de la mayoría de los países. Como los métodos de PCR actualmente enumerados en el Manual Acuático de la OIE no pueden distinguir entre virus vivos y EVEs, a menos que se lleve a cabo la secuenciación del IHHNV, creemos que la OIE debe tomar medidas para corregir este problema.
En conclusión, ¿deberían los camaroneros de Ecuador preocuparse por el impacto del IHHNV en la producción de sus granjas? No lo creemos. Además, la OIE debería eliminar el IHHNV de la lista de patógenos notificables de los camarones peneidos o, al menos, revisar los métodos de PCR actualmente enumerados para el IHHNV en el Manual Acuático.
Ahora que Ud. ha terminado de leer este artículo...
… esperamos que considere apoyar nuestra misión de documentar la evolución de la industria acuícola mundial y compartir el conocimiento expansivo de nuestra amplia red de colaboradores cada semana.
Al convertirse en miembro de la Global Seafood Alliance, se asegura de que todo el trabajo pre-competitivo que realizamos a través de beneficios, recursos y eventos para miembros pueda continuar. La membresía individual cuesta solo $50 al año.
¿No es miembro de la GSA? Únase a nosotros.
Authors
-
James A. Brock, D.V.M, retd.
Corresponding author
95-430 Kamahana Place, Mililani, Hawaii USA[109,111,99,46,114,114,46,105,105,97,119,97,104,64,49,48,48,106,107,99,111,114,98]
-
Pablo Intriago, Ph.D.
South Florida Farming Corp., Southwest Ranches, Florida USA; and Empagran, ABA Division Balanceado, Guayaquil, Ecuador
-
Andres Medina, M.Sc.
Empagran S.A. Semacua Division Laboratories, Guayaquil, Ecuador
Tagged With
Related Posts

Health & Welfare
AHPND es una enfermedad crónica del camarón blanco del Pacífico de América Latina
Hay una nueva fase de infección para la enfermedad de Necrosis Hepatopancreática Aguda en las granjas camaroneras de América Latina, en contraste con las observaciones del sudeste asiático.

Health & Welfare
Cómo los niveles de salinidad afectan las tasas de infección por EHP en el camarón blanco del Pacífico
Una infección por EHP puede ocurrir a una salinidad tan baja como 2 ppt, pero la prevalencia y la gravedad de la infección por EHP es mayor a una salinidad de 30 ppt.

Aquafeeds
Efectos de varios atrayentes de piensos en juveniles de camarón blanco del Pacífico
La ganancia de peso y el consumo de alimento del camarón blanco del Pacífico aumentaron con la inclusión dietética de ciertos atrayentes alimenticios y disminuyeron con otros.

Health & Welfare
EHP es un factor de riesgo para otras enfermedades del camarón
Desafíos de laboratorio y un estudio caso-control se utilizaron para determinar los efectos de la infección por EHP en dos enfermedades de Vibrio: necrosis hepatopancreática aguda (AHPND) y necrosis hepatopancreática séptica (SHPN).



