Se observó una expansión significativa de familias de genes en el genoma relacionados con la locomoción, la visión y la transmisión neuronal

Un genoma es toda la información genética de un organismo. Un genoma de referencia de alta calidad es crucial para la mejora genética de las especies y la exploración de los mecanismos moleculares subyacentes a importantes procesos biológicos. Sin embargo, los genomas del camarón contienen numerosas secuencias repetitivas, lo que hace que su ensamblaje sea un desafío. Las tecnologías de secuenciación de tercera generación desarrolladas recientemente han ayudado a aliviar las dificultades de ensamblaje causadas por la heterocigosidad y las secuencias repetitivas en los genomas.
Recientemente se han ensamblado los genomas de varias especies de camarón de importancia comercial, incluido el camarón tigre negro (Penaeus monodon), el camarón blanco chino (Fenneropenaeus chinensis), el camarón Kuruma (Marsupenaeus japonicus) y otros, utilizando estas nuevas tecnologías de secuenciación de tercera generación. Sin embargo, en comparación con otras especies, la disponibilidad de genomas de camarón aún es limitada.
Previamente se ha generado un borrador del genoma de L. vannamei, aunque está muy fragmentado y no está construido a nivel cromosómico. La falta de un genoma de alta calidad impide futuras investigaciones genéticas sobre L. vannamei.
Este artículo – resumido de la publicación original (Peng, M. et al. 2023. A high-quality genome assembly of the Pacific white shrimp (Litopenaeus vannamei) provides insights into its evolution and adaptation. Aquaculture Reports Volume 33, December 2023, 101859) – informa sobre un estudio para generar un ensamblaje genómico de alta calidad de L. vannamei y proporcionar nueva información genómica crítica para la cría de camarones y las investigaciones de esta especie de camarón.
Configuración del estudio
Un ensamblaje de novo se refiere al ensamblaje del genoma de un nuevo genoma desde cero sin la ayuda de datos genómicos de referencia. Para la secuenciación y el ensamblaje del genoma, se obtuvieron muestras de L. vannamei del Centro de mejoramiento genético de camarones de la Academia de Ciencias Pesqueras de Guangxi (Nanning, China). El ADN genómico se extrajo del tejido muscular y de las bibliotecas genómicas utilizando un kit comercial (kit SMRTbell, PacBio, EE. UU.) de acuerdo con el protocolo del fabricante y se secuenció en la plataforma PacBio Sequel. Luego, este ADN fue procesado y analizado en detalle.
Para obtener información detallada sobre la secuenciación y ensamblaje del genoma de L. vannamei; anotación genética; anotación de secuencias repetidas; secuenciación transcriptómica de diversos tejidos; y análisis evolutivo, consultar la publicación original.
Resultados y discusión
L. vannamei ha recibido una amplia atención de la investigación debido a su enorme valor económico. Anteriormente se publicó un borrador del genoma de esta especie, aunque este ensamblaje genómico anterior permanece relativamente fragmentado e incompleto, lo que limita su aplicabilidad como referencia para el mejoramiento y la investigación.
Para obtener un genoma resuelto de L. vannamei, utilizamos tecnologías novedosas en este estudio para generar un ensamblaje de novo del genoma de alta calidad, con una longitud total de 1869 megabases (Mbp; megabases o millones de pares de bases, es una de las formas utilizadas para medir el tamaño de un genoma) y otras características mejoradas. Estas métricas mejoran considerablemente el ensamblaje del genoma incompleto publicado anteriormente.
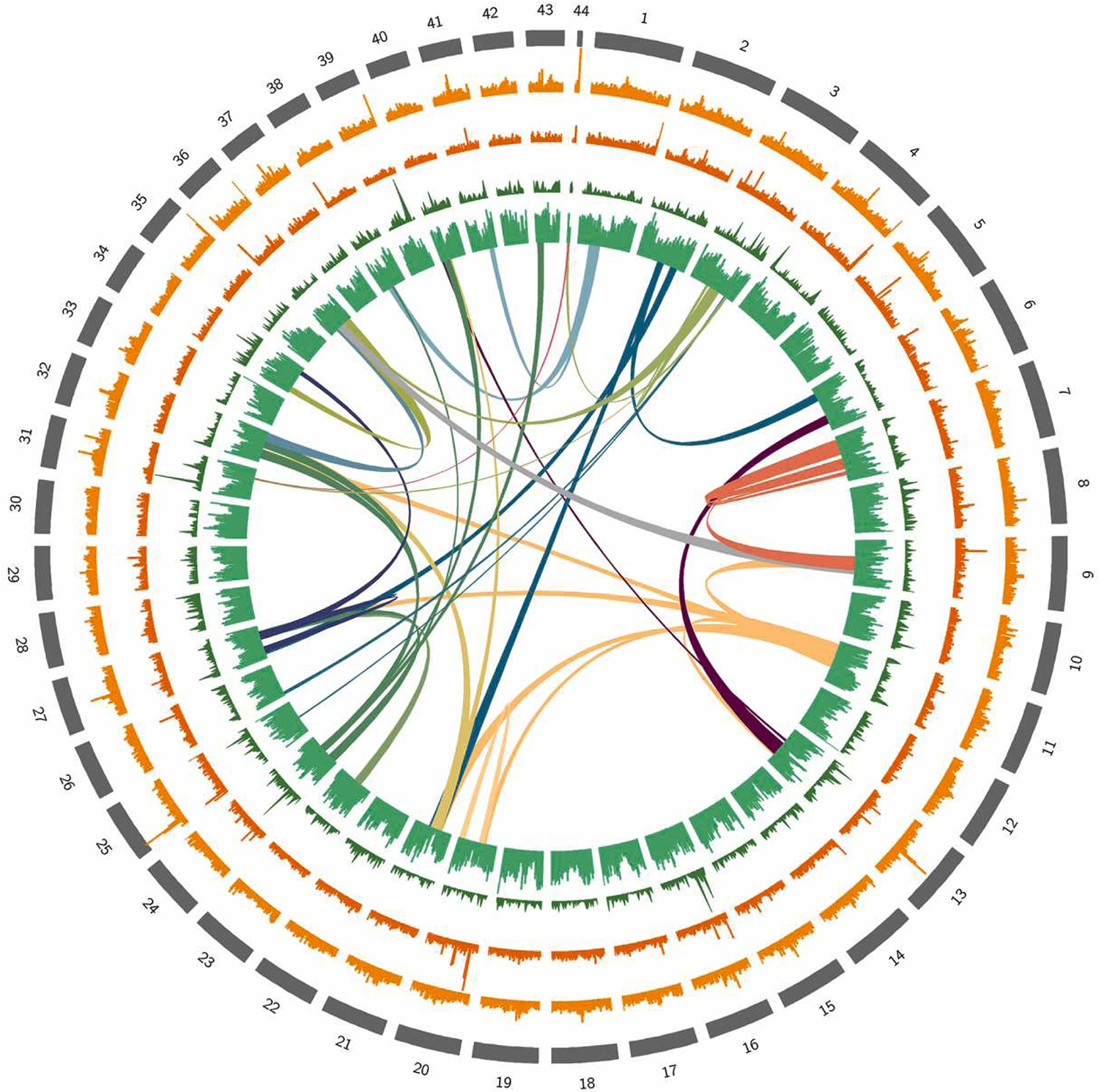
Este ensamblaje genómico mejorado proporciona una representación más completa y precisa del genoma de L. vannamei, lo que permite conocer mejor su composición genética y sus mecanismos reguladores. También abre nuevas oportunidades para la investigación y las aplicaciones. Los investigadores ahora pueden explorar el contenido genético, la evolución y los aspectos funcionales de L. vannamei con mayor detalle, incluidas investigaciones sobre rasgos clave como el crecimiento, la reproducción, la resistencia a enfermedades y la adaptación ambiental.
Además, este ensamblaje genómico de alta calidad sirve como una referencia invaluable para futuros estudios sobre la genómica de L. vannamei, permitiendo la identificación y anotación de genes y elementos reguladores previamente desconocidos y facilitando estudios de genómica comparativa para comprender las relaciones evolutivas y la base genética de la diversificación fenotípica dentro de la familia de los camarones.
Si bien investigaciones anteriores han sugerido que el recuento de cromosomas haploides (un solo conjunto) de L. vannamei es 44, puede ser necesaria más investigación para determinar con precisión el número exacto de cromosomas en esta especie.
En cuanto a la anotación de secuencias repetitivas (patrones cortos o largos de ácidos nucleicos – ADN o ARN – que se producen en múltiples copias a lo largo del genoma), nuestros resultados mostraron una proporción menor en comparación con un estudio anterior. Esta variación podría deberse a la utilización de técnicas mejoradas de ensamblaje del genoma en nuestra investigación o a la posibilidad de que falten repeticiones en nuestro ensamblaje.
Las secuencias repetitivas desempeñan un papel crucial en la estructura y función del genoma, ya que se sabe que regulan la expresión genética en F. chinensis y L. vannamei, impulsando la evolución adaptativa, como la capacidad osmorreguladora variable de estos camarones bajo estrés de baja salinidad. Los estudios futuros deberían centrarse en abordar las limitaciones de nuestro ensamblaje para mejorar nuestra comprensión del contenido repetitivo dentro del genoma de L. vannamei.
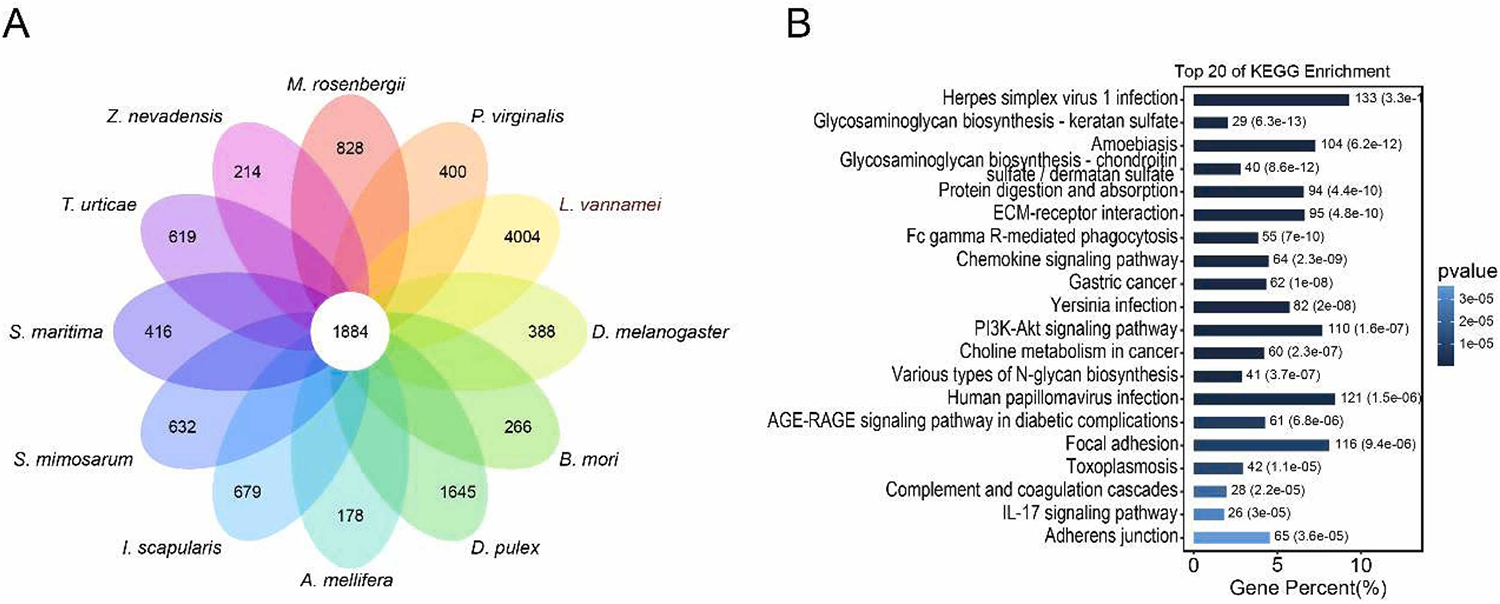
La expansión o contracción de familias de genes son mecanismos clave en la evolución adaptativa de los organismos. Observamos una expansión significativa de algunas familias de genes en el genoma de L. vannamei. Las familias de genes ampliadas incluyeron algunas que participan principalmente en funciones de movimiento, además de otras familias de genes involucradas en funciones de transmisión visual y neuronal. La expansión significativa de estas familias de genes podría conducir a una mayor capacidad visual, de velocidad neuronal y de movimiento de L. vannamei, lo que tal vez permitiría su adaptación para escapar de la depredación en hábitats bentónicos oscuros. El abundante repertorio de genes en el genoma de L. vannamei relacionados con la transmisión visual, neuronal y el movimiento puede explicar sus adaptaciones bentónicas.
También construimos un árbol filogenético que involucra a L. vannamei y otras 11 especies de artrópodos. Nuestros hallazgos sugieren que L. vannamei está estrechamente relacionado con otros crustáceos como el cangrejo jaspeado (Procambarus virginalis) y el langostino gigante de agua dulce (Macrobrachium rosenbergii) y respaldan estudios previos. Sin embargo, cabe destacar que la pulga de agua (Daphnia pulex), otro crustáceo, no se agrupaba con L. vannamei, y esto puede deberse a su pequeño tamaño de genoma de sólo 200 Mbp, lo que da lugar a diferencias genéticas significativas en comparación con L. vannamei, con un tamaño de genoma mucho mayor. Este resultado contradice los resultados de investigaciones anteriores y se requiere más investigación en estudios futuros.
Perspectivas
Este estudio documentó la generación de un ensamblaje de novo de alta calidad del genoma de L. vannamei, abordando las limitaciones de ensamblajes fragmentados anteriores. Nuestro producto refinado no solo proporciona una representación más precisa del genoma de L. vannamei, sino que también ofrece nuevas vías para la genómica funcional, programas de mejoramiento y estudios evolutivos comparativos en esta especie económicamente significativa.
Ahora que Ud. ha llegado al final del artículo...
… por favor considere apoyar la misión de la GSA de promover prácticas responsables de productos del mar a través de la educación, la defensa y las garantías de terceros. El Advocate tiene como objetivo documentar la evolución de las prácticas responsables de productos del mar y compartir el amplio conocimiento de nuestra amplia red de colaboradores.
Al hacerse miembro de la Global Seafood Alliance, está asegurando de que todo el trabajo pre-competitivo que hacemos a través de los beneficios, recursos y eventos para miembros pueden continuar. La membresía individual cuesta solo $50 al año.
¿No es miembro de la GSA? Únase a nosotros.
Author
-
Dr. Yongzhen Zhao
Autor de correspondencia
Guangxi Key Laboratory of Aquatic Genetic Breeding and Healthy Aquaculture, Guangxi Academy of Fishery Sciences, Nanning 530021, China[32,109,111,99,46,108,105,97,109,116,111,104,64,111,97,104,122,110,101,104,122,103,110,111,121]
Tagged With
Related Posts

Health & Welfare
Calidad, supervivencia de la progenie de L. vannamei de hembras sometidas a ablación y a no-ablación
Evaluación de la calidad y supervivencia de postlarvas de camarón blanco del Pacífico de reproductores hembras no sometidas a ablación, una estrategia holística de bioseguridad y gestión.

Health & Welfare
¿Protegen las dietas suplementadas con fitobióticos al camarón blanco del Pacífico contra AHPND?
Las dietas de camarones suplementadas con fitobióticos y aplicadas en períodos críticos podrían mitigar el impacto de AHPND y otras patologías.

Health & Welfare
La comunidad microbiana de camarones supervivientes tratados con probióticos se somete a prueba de desafío de AHPND
Evaluando la aplicación práctica de probióticos para manejar patógenos que causan altas tasas de mortalidad en el cultivo de camarón en todo el mundo.

Health & Welfare
Indonesian producer turns to genetic technology to enhance its shrimp breeding programs
How Indonesian company Prima Larvae Bali is using genetic technology and a partnership with CAT to elevate its shrimp breeding program.



